Quand on s'intéresse à la complexité des algorithmes il est intéressant de pouvoir confronter la théorie avec la pratique en vérifiant quel est le temps d'exécution réel d'un programme et comment ce temps est réparti entre les différentes opérations. Il existe des outils de profilage de code qui permettent d'analyser le coût de chaque ligne de code dans un programme (coût du temps de calcul du processeur ou en mémoire). Scilab possède quelques fonctionnalités dédiées à ce sujet, que j'avais documenté il y a plus de 10 ans , mais qui ont été complètement remanié. Le développement de Scilab semblant retrouver de l'activité, je me suis donc intéressé aux capacités des nouvelles fonctions de profilage et à l'exploitation graphique des résultats.
Les commandes de profilage Scilab
scilab propose 3 commande de profilage
- ProfileEnable et ProfileDisable pour ajouter/enlever les commandes de profilage à la fonction
- profileGetInfo() pour récupérer les résultats d'analyse
profileEnable(fct) // ajoute les commande de profilage à fct fct(donnes_test) // exécution de fct avec des données de test profile = profileGetInfo() // récupération des informations sur l'exécution de fct profileDisable(fct) // retire les commandes de profilage de fctle résultat profile est une structure de donnée en apparence compliquée quand on l'affiche dans la console de scilab :
profile =
profile(1)
"ProfilerStatistics"
"FunctionTable"
"FunctionCoverage"
"LineCoverage"
profile(2)
FunctionName = "nom_fct"
FileName: [1x1 string]
FirstLine = 35
LibraryName = "script"
ParentIndex = 0
profile(3)
NumCalls = uint64(0)
TotalTime = 0
InstructionsCount = uint64([0,0])
BranchesCount = uint64([0,0])
PathsCount = uint64(0)
profile(4)
(1) : [9x2 constant]mais en fait il n'y a que deux informations qui vont me servir ensuite :
- profile.FunctionTable.FunctionName ou profile(2).FunctionName contient le nom (chaîne de caractère) de la fonction à tester
- profile.LineCoverage(1) ou profile(4)(1) contient un tableau avec 2 colonne contenant :
- un code pour chaque ligne du programme (-1 pour des mots clé ne générant pas d'exécution, code >0 pour les autres lignes)
- le temps CPU utilisé par chaque ligne lors du test
Utilisation sur un exemple
je vais prendre l'exemple d'un programme effectuant un tri fusion :
function L=tri_fusion(L) n=length(L) // longueur de L if n>1 then m=int(n/2) // couper la liste en deux L1=tri_fusion(L(1:m)) L2=tri_fusion(L(m+1:$)) L=fusionner(L1,L2) end endfunction function L=fusionner(L1, L2) n1=length(L1) n2=length(L2) n=n1+n2 L=zeros(1,n) L1($+1)=%inf L2($+1)=%inf i1=1,i2=1,k=0 while k<n k=k+1 if L1(i1)<L2(i2) then L(k)=L1(i1),i1=i1+1 else L(k)=L2(i2),i2=i2+1 end end endfunction
Pour tester cette fonction il faut donc créer une liste de valeurs aléatoires (avec grand) puis lui appliquer tri_fusion en ayant activé le profilage comme ci-dessous :
profileEnable(tri_fusion) // ajoute les commande de profilage à tri_fusionon peut ensuite examiner les résultats d'analyse contenus dans profile.LineCoverage(1) :
n=1000;L=grand(1,"prm",[1:n]); // données pour les tests tri_fusion(L) // exécution de tri_fusion avec des données de test profile = profileGetInfo() // récupération des informations d'exécution de tri_fusion profileDisable(tri_fusion) // retire les commandes de profilage de tri_fusion
-->profile.LineCoverage(1)
ans =
-1. 0.
1999. 0.0006279
1999. 0.0001537
999. 0.0064534
999. 0.0010096
999. 0.0009101
999. 0.0177951
-1. 0.
-1. 0. c'est la deuxième colonne qui contient les temps CPU correspondant à chaque ligne de code. On peut aussi récupérer les lignes de code de la fonction pour les mettre en correspondance avec les résultats d'analyse. Pour cela on récupère le code de la fonction avec string :
-->[in,out,text]=string(tri_fusion)
in =
"L"
out =
"L"
text =
" "
"n = length(L),// longueur de L;"
"if (n > 1) then"
" m = int(n / 2),// couper la liste en deux;"
" L1 = tri_fusion(L((1:m)))"
" L2 = tri_fusion(L((m + 1:$)))"
" L = fusionner(L1, L2)"
"end"
" " vous pouvez voir qu'on retrouve les 9 lignes de code de tri_fusion sauf les deux lignes correspondant aux 2 mots-clés function et endfunction. Attention a ne pas écrire de commande sur les lignes contenant des mot-clés comme if ... then ou else .. car ces commandes sont déplacées par la fonction string et le résultat ne correspond plus aux lignes analysées par les fonctions de profilage!
Si on a fait attention à cela on peut ensuite utiliser la deuxième colonne de profile.LineCoverage(1) pour calculer le pourcentage de temps CPU consommé par chaque ligne :
tps_CPU=profile.LineCoverage(1)(:,2)
tps_CPU =
0.
0.0006279
0.0001537
0.0064534
0.0010096
0.0009101
0.0177951
0.
0.
-->tps_CPU=tps_CPU/sum(tps_CPU)
tps_CPU =
0.
0.0233008
0.0057025
0.2394597
0.0374607
0.0337694
0.6603070
0.
0. Mise en forme graphique
on peut utiliser ces données de profilage pour réaliser un diagramme en bâton représentant l'importance de chaque ligne. Voici un exemple de mise en forme graphiques de ces données en utilisant la fonction barh pour créer un diagramme en bâtons horizontal ajout des lignes de code grâce à string et xstring : :
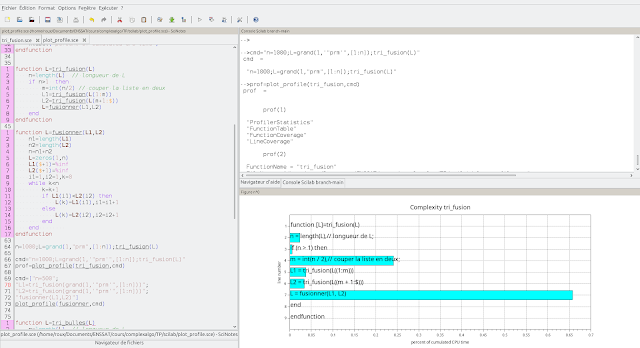
function prof=plot_profile(fct, cmd) // fct= fonction scilab à tester //cmd = commandes de test (chaîne de caractères) // profilage de la fonction profileEnable(fct) execstr(cmd); // test de la fonction prof = profileGetInfo() profileDisable(fct) fct_name=prof(2).FunctionName // nom de la fonction tab=prof.LineCoverage(1);// récupération des données tps_CPU=tab(:,2)/sum(tab(:,2)); // % du tps CPU clf() // ouverture fentregraphique nl=size(tps_CPU,1) // nombre de lignes de la fonction barh(1:nl,tps_CPU($:-1:1),0.8,"cyan") //histogramme horizontal // extraction des lignes de code de la fonction [out,in,text]=string(fct) text(1)="function ["+strcat(out,",")+"]="+fct_name+"("+strcat(in,",")+")" text(nl)="endfunction" // ajout des lignes de code dans le graphe for k=1:nl xstring(0,k-0.3,text(nl+1-k)) e=gce(); e.font_size=4; e.font_foreground=1; end // titre et modification des labels xtitle("Complexity "+fct_name) xgrid(1) gca().title.font_size=4 gca().y_ticks.labels=string([nl:-1:1]') ylabel("line number") xlabel("percent of cumulated CPU time") endfunctionj'ai décidé de fournir à la fonction plot_profile la commande pour faire les tests (variable cmd) sous forme d'une chaîne de caractères pour simplifier mon utilisation. voici le résultat obtenu pour la fonction tri_fusion :
on peut voir que c'est la fonction fusionner qui effectue la tâche principale dans ce tri, fonction que l'on peut ensuite analyser de la même manière :
cmd=["n=500"; "L1=tri_fusion(grand(1,'"prm'",[1:n]))"; "L2=tri_fusion(grand(1,'"prm'",[1:n]))"; "fusionner(L1,L2)"] plot_profile(fusionner,cmd)là ce sont les opération de la boucle while qui prennent le plus de temps CPU, puisqu'elle sont répétées n fois.


Aucun commentaire:
Enregistrer un commentaire
Pour écrire des formules mathématiques vous pouvez utiliser la syntaxe latex en mettant vos formules entre des "dollars" $ \$....\$ $ par exemple :
- $\sum_{n=1}^\infty {1\over n^2}={\pi^2\over 6}$ s'obtient avec \sum_{n=1}^\infty {1\over n^2}={\pi^2\over 6}
- $\mathbb R$ s'obtient avec {\mathbb R} et $\mathcal D$ s'obtient avec {\mathcal D}
- pour les crochets $\langle .,. \rangle$ dans les commentaires utilisez \langle .,. \rangle
vous pouvez écrire du html dans les commentaires :
- italique <i> ... </i> gras <b> ... </b>
- lien <a href="http://adresse "> .... </a>